.
Les réseaux de neurones et les réseaux alimentaires sont d’autres exemples d’origine biologique. Mais il y a des réseaux sociaux d’acteurs ou de mathématiciens, des réseaux d’information comme les réseaux des citations, l’Internet ou le World Wide Web et des réseaux technologiques comme les réseaux des centrales électriques d’un pays ou l’Internet2 dont l’origine n’est plus biologique. Tous ces réseaux ont quelques propriétés communes comme l’existence de « courts chemins » en moyenne 4La distance moyenne entre deux noeuds est donnée par \[l = 2 \Sigma \frac{d_{ij}}{n (n+1)}\] où \(n\) est le nombre de noeuds et \(d_{ij}\) est la distance minimale entre deux noeuds. Dans un réseau du type « monde petit » cette distance est petite. – l’effet « small world » ou « du monde petit » 5 Pensons à Hollywood où « presque tout le monde » a travaillé avec Kevin Bacon –, un taux élevé d’agrégation ou « clustering » 6Le {taux d’agrégation} ou {« clustering »} d’un réseau est la moyenne des quantités \(2e_v/k_v(k_v-1)\) associées aux noeuds \(v\) où \(e_v\) représente le nombre d’arêtes entre voisins de \(v\) et \(k_v\) le degré de \(v\). Quand on dit qu’un réseau réel montre un degré d’agrégation élevé, c’est par comparaison avec un réseau aléatoire. – de manière que les voisins d’un noeud ont toujours d’autres voisins – ou une distribution du degré de noeuds selon une loi de puissance 7D’un point de vue déterministe, on peut dire que le degré des noeuds d’un réseau suit une {loi de puissance} lorsque \(p(k) =\) nombre de noeuds de degré \(k = c k^{-\gamma}\) où \(c\) et \(\gamma\) sont des constantes. Si on pense au nombre de noeuds comme une variable aléatoire, on dira que la distribution du degré des noeuds est gouvernée par une {loi de puissance} si la fraction du nombre de noeuds de degré \(k\) s’approche de la quantité \(c k^{-\gamma}\) lorsque \(k\) devient de plus en plus grand. On écrit alors \(p(k) \sim c k^{-\gamma} \). Les graphes aléatoires suivent une distribution de Poisson \(p(k) \sim e^{-\gamma}\frac{\gamma^k}{k!}\).– avec beaucoup de noeuds faiblement connectés et peu de noeuds fortement connectés –.
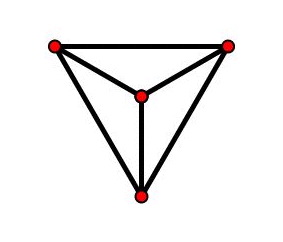
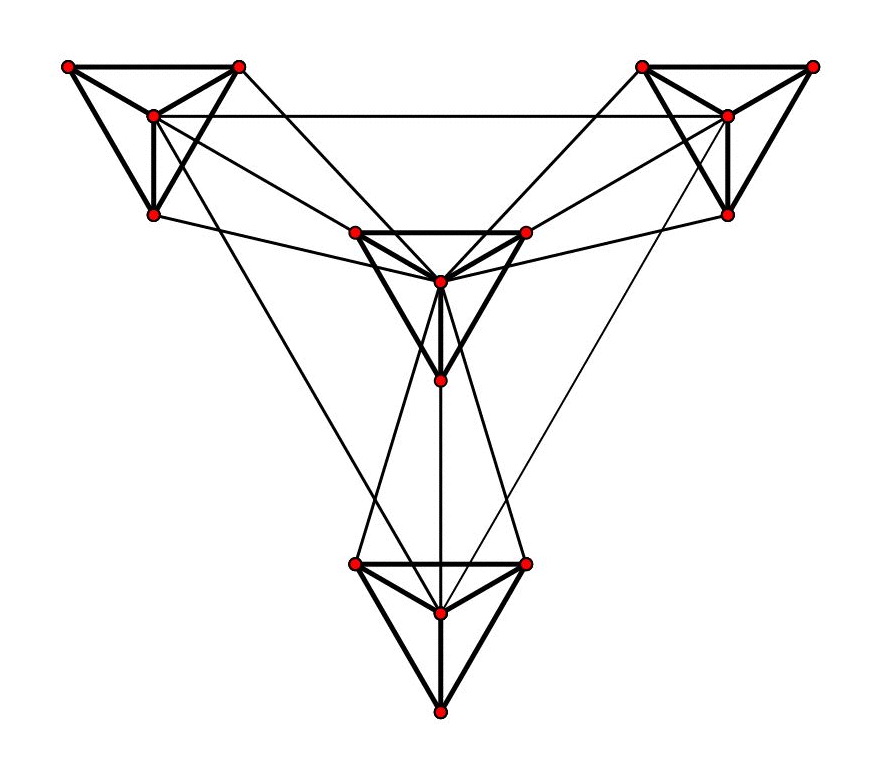
En 2002, l’équipe du professeur Uri Alon du Weizmann Institut of Science observa que ces réseaux contiennent des petits sous-graphes surreprésentés, qu’ils appelèrent motifs 8R. Milo, S. Shen-Orr, S. Itzkovitz, N. Kashtan, D. Chklovskii, U. Alon, Motifs : Simple Building Blocks of Complex Networks. Science 298 (2002), 824-827. Ces sous-graphes apparaissent dans les réseaux avec des fréquences plus élevées que celles que l’on trouve dans des réseaux aléatoires ayant la même distribution de noeuds. Ils montrent des hauts taux de conservation entre des organismes différents. Voici les motifs surreprésentés trouvés dans le réseau de neurones du nématode Caenorhabditis elegans (252 neurones et 509 connexions) :
.
L’idée d’une fonction biologique attachée aux motifs du réseau de neurones de ce petit ver, qui deviennent ainsi des modules fonctionnels, est très intéressante. Mais comme il a été remarqué par d’autres auteurs, il faut faire attention aux faux positifs dérivés de l’algorithme de recâblage utilisé pour engendrer les réseaux aléatoires et au fait que certains réseaux – comme celui de neurones du ver Caenorhabditis elegans – ont une structure spatiale qui favorise l’agrégation de neurones de façon locale 9Y. Artzy-Randrup, S. J. Fleishman, N Ben-Tal, L. Stone, Comment on « Network Motifs : Simple Building Blocks of Complex Networks » and « Superfamilies of Evolved and Designed Networks ». Science, 305 (2004), 1107. Dans ce travail, les auteurs construisent un « réseau de jouet » à partir des noeuds d’une grille composée de 30×30 carrés où la probabilité de joindre deux noeuds par un arc décroît avec la distance suivant une distribution gaussienne. Les motifs présents dans le réseau de neurones du ver C. elegans sont aussi surreprésentés dans ce réseau aléatoire.. Néanmoins l’idée d’une modularité propre de certains réseaux biologiques (où l’agrégation de modules fonctionnels simples – très conservés entre les différentes espèces – amène à des larges et complexes structures, chevauchées et inséparables, caractéristiques de chacune des espèces) reste très suggestive au moins pour un mathématicien 10De la même manière que l’arbre de Kenyon évoque pour moi des morceaux de Kafka et de Bach, la lecture de l’épilogue du livre An introduction to systems biology de Uri Alon (publié par Chapman & Hall à Boca Raton en 2007) sur le rôle de la modularité et de la probabilité dans les systèmes biologiques me fait penser inévitablement à ses compagnons – les arbres indistinguables de l’arbre de Kenyon – et aux pavages de Penrose..
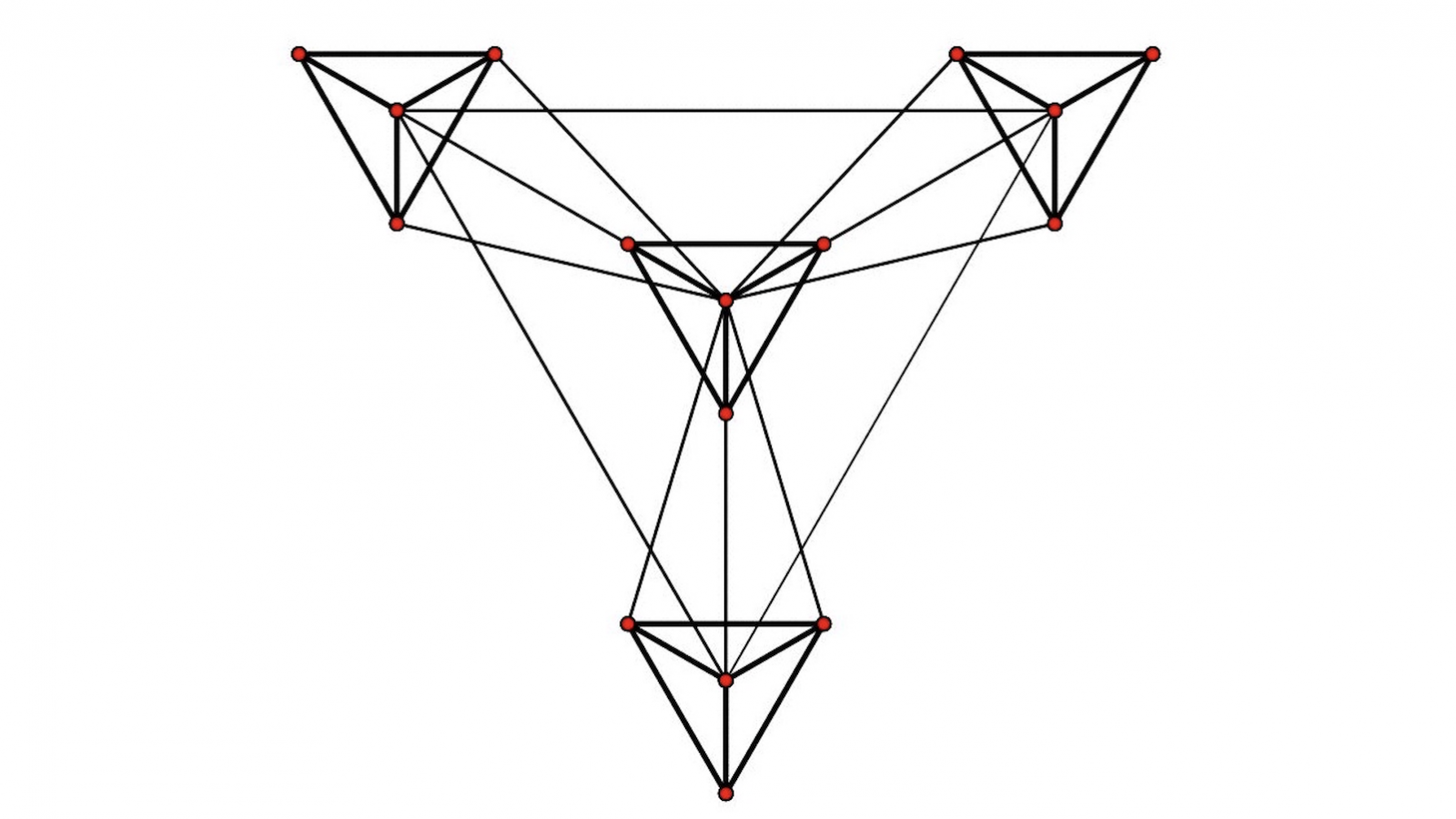
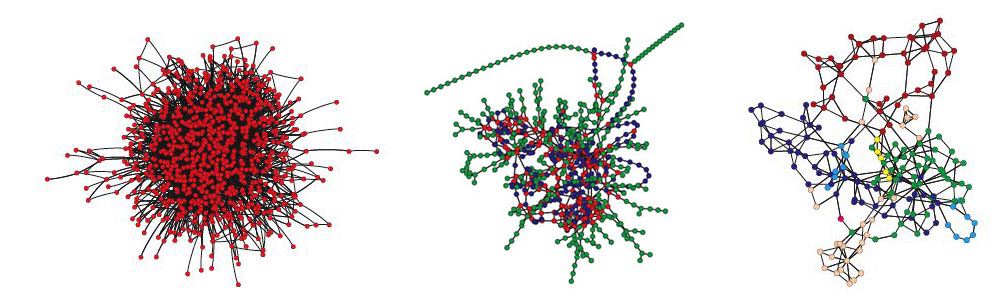
Des différent types de modèles de réseaux cherchent à capturer les propriétés essentielles des réseaux du monde réel. Le modèle aléatoire de Erdös-Rényi qu’on obtient en reliant chaque paire de noeuds avec une probabilité fixe \(0 \leq p \leq 1\), possède la propriété de « courts chemins » propre des « mondes petits », mais pas les autres propriétés. Le modèle de Watts-Strogatz permet d’augmenter le taux d’agrégation, mais la distribution du degré de noeuds reste poissonnienne. Pour construire un modèle dont la distribution du degré des noeuds soit gouvernée par une loi de puissance, ont peut utiliser un algorithme, appelé modèle de Barabási-Albert, qui consiste à ajouter un noeud et à le relier aux noeuds existants (énumérés \(i=1,…n\)) avec une probabilité \(p_i = k_i / \sum_{i=1}^n k_i\), dite d’attachement préférentiel, proportionnelle au degré \(k_i\) de chaque noeud \(i\).Ce sont des modèles, dits sans échelle, très robustes ou insensibles aux erreurs aléatoires, mais très vulnérables aux attaques sur les noeuds de haut degré ou « hubs ». Mais dans ce modèle le taux d’agrégation tend vers 0 lorsque la taille du réseau augmente, ce qui ne se correspond pas avec l’observation. L’idée de réseau hiérarchique, introduite par E. Ravasz de l’équipe de A. L. Barabási, cherche à éliminer ce problème 11Ravasz, A. L. Somera, D. A. Mongru, Z. N. Oltvai, A. L. Barabási, Hierarchical organization of modularity in Metabolic networks. Science, 297 (2002), 1551-1555.. Il s’agit de combiner de manière itérative des petites agrégations de motifs. Voici un exemple réseau hiérarchique décrit dans l’article de Ravasz où le centre d’un « module clef » est connecté aux « noeuds périphériques » (appartenant aux sous-modules périphériques) de trois « modules périphériques » et les centres des ces modules sont interconnectés.
.
.

.
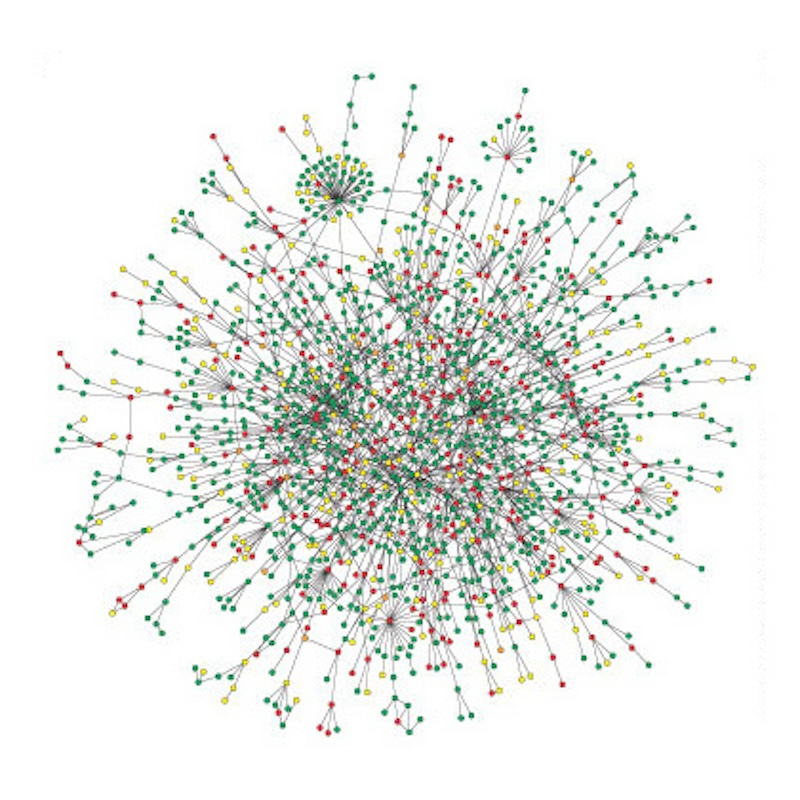
Remarquons que tout sous-graphe fini peut être retrouvé à distance bornée de n’importe quel noeud. Mais la nature répétitive de ce réseau est moins rigide que celle du pavage de Kepler-Penrose ou de l’arbre de Kenyon où tout sous-graphe est retrouvé de façon fidèle, c’est-à-dire en tenant compte des arêtes présentes et absentes. Dans les réseaux hiérarchiques, le taux d’agrégation s’approche d’une constante – qui vaut 0,606 pour l’exemple ci-dessus – indépendante du nombre des noeuds, mais la fonction c(k) qui mesure la taux d’agrégation des noeuds de degré k suit une loi de puissance – \(c(k) \sim k^{-1}\) pour l’exemple –. Le réseau métabolique du bacille Escherichia Coli est modelé par Ravasz et ses coauteurs en utilisant ce type de réseau. Grâce à un procédé de réduction – illustré dans la figure ci-dessous –, ils se ramènent à un réseau modulaire, puis ils se servent de l’invariance d’échelle pour affirmer leur nature hiérarchique
.
Il y a d’autres auteurs comme John Doyle qui critiquent cette approche et qui proposent d’autres modèles pour expliquer l’architecture de certains réseaux – souvent liés au génie informatique et industriel – n’ayant pas les propriétés des réseaux hiérarchiques. La loi d’échelle pour le taux d’agrégation est effectivement une condition nécessaire, mais pas suffisante pour l’existence d’une structure hiérarchique. Le modèle HOT (« Highly Optimized Tolerance » ou « Heuristic Optimized Tradeoff ») proposé par Doyle – qui cherche à expliquer la manière d’optimiser les performances sous contraintes technologiques ou economiques – se veut opposé à celui de Ravasz, auto-dissimilaire et à échelle riche.
Mais l’existence d’un « noyau » dans ce modèle est semblable à celle du noyau dans le pavage de Dürer : bien qu’on n’ait plus de structure répétitive ou auto-similaire (en un sens à préciser), cela ne signifie pas que le réseau ne garde pas une sorte de répétitivité ou d’auto-similarité consubstantielle à la notion de modularité.
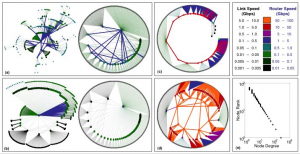
Voici les différents type de réseaux représentés dans la figure : (a) Modèle de Barabási-Albert (b) Modèle sans échelle (c) Réseau mauvais (d) Réseau HOT
Comme j’ai déjà dit au début, il s’agit d’idées qui ne sont pas totalement établies. Mais, discutées ou non, elles dessinent à mon avis une belle esquisse du rôle de la biologie dans les maths à venir 12Je ne pense pas seulement aux « mathematical sciences », mais aux « core mathematics » pour répéter les formules utilisées récemment par F. Quinn dans les Notices of the American Mathematical Society.. Le regard sur les mathématiques du début du XXe siècle, alors qu’on commémore le centenaire de la mort d’Henri Poincaré, peut nous donner une idée la taille du défi et ses dangers.

Il est possible d’utiliser des commandes LaTeX pour rédiger des commentaires — mais nous ne recommandons pas d’en abuser ! Les formules mathématiques doivent être composées avec les balises .
Par exemple, on pourra écrire que sont les deux solutions complexes de l’équation .
Si vous souhaitez ajouter une figure ou déposer un fichier ou pour toute autre question, merci de vous adresser au secrétariat.